




 Artículos originales> Expertos del Mundo>
Artículos originales> Expertos del Mundo> Enviar correspondencia a:
Enviar correspondencia a: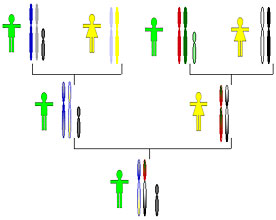
Figura 1. Comparación de la herencia directa varón-varón del cromosoma Y. Los demás cromosomas se "mezclan" cuando pasan de una generación a la siguiente, una complicación para establecer el origen de los individuos analizando estos cromosomas.Siguiendo el origen los CY humanos, llegaríamos a un ancestro paterno único análogo al "Adán" bíblico. Sin embargo, cuando se transmite el CY de generación en generación, se van acumulando cambios o mutaciones en el ADN a través del tiempo, a los que se les llama marcadores moleculares en pruebas de identidad, y que a su vez constituyen un registro claro de nuestro pasado. Durante los últimos años se ha extendido el análisis del CY, tanto en las ciencias forenses como en la antropología, el objetivo de este trabajo es dar a conocer las particularidades de estos estudios. Cabe mencionar que existe una región en los extremos del CY que sí se "mezcla" con el otro cromosoma sexual (X), pero que pierde interés para estudios antropológicos. Nos referiremos en este trabajo exclusivamente a la parte del CY que no recombina.II. Marcadores del cromosoma Y (CY)
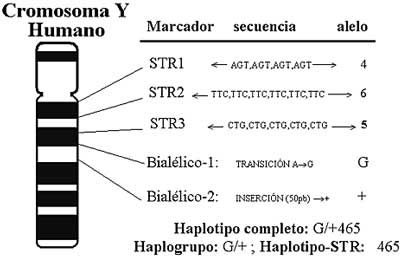
Figura 2. Determinación de un haplogrupo-haplotipo de un individuo al analizar marcadores bialélicos y STRs del cromosoma Y humano, respectivamente.III. El cromosoma Y en ciencias forenses
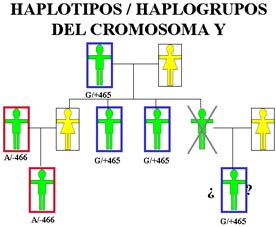
Figura 3. Establecimiento de paternidad en un caso donde el supuesto padre no está disponible mediante el análisis de marcadores del cromosoma Y.Es importante considerar que los haplotipos del CY son compartidos por todos los parientes vía paterna, que constituyen linajes paternos y que suelen agruparse dentro de las poblaciones de formas muy particulares de acuerdo con sus historias. Por consiguiente, es necesario considerar que en una prueba forense o de paternidad puede existir un número indeterminado de varones con el mismo haplotipo, además de los parientes por línea paterna del sujeto implicado [12, 13]. Por esta razón, es necesario establecer las frecuencias poblacionales, no únicamente de los alelos, sino de los haplotipos del CY en cada población donde se quiera emplear esta tecnología, como ya se ha reportado para varias poblaciones del mundo [14, 15]. De manera que, aunque los haplotipos del cromosoma Y pueden utilizarse confiablemente para realizar exclusiones de paternidad o culpabilidad, se recomienda usar otros marcadores moleculares localizados en cromosomas no-sexuales para llevar a cabo las inclusiones [12, 13]. Cabe recordar que una exclusión implica el descartar como padre, o como fuente de una muestra forense, a un hombre falsamente implicado en el caso. IV. El cromosoma Y en antropología
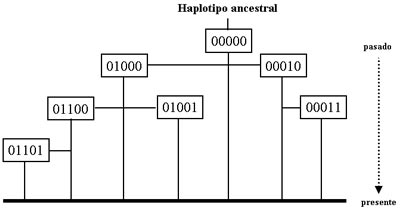
Figura 4. Establecimiento de las relaciones evolutivas entre haplogrupos del CY (marcadores bialélicos) observados en una población, mediante el método de parsimonia. El estado ancestral se establece por comparación con antropoides superiores (bonobos, chimpancés, orangutanes, etc.) .Muchos marcadores bialélicos (haplogrupos) pueden relacionarse con un lugar geográfico específico, normalmente el lugar donde se originó y/o expandió la mutación, por lo que son una valiosa herramienta para saber el origen paterno del varón que lo porta (caucásico, asiático, amerindio, africano, etc.) [4]. La relación haplogrupo-geografía ha permitido confirmar el origen del hombre en África: los haplogrupos más antiguos están en este continente y todos los humanos tenemos algunas mutaciones africanas [16-18]. De igual forma, marcadores bialélicos del CY han permitido reconstruir el proceso de dispersión del hombre desde su salida de África hasta poblar todo el globo terráqueo [8, 18]. ¿Por qué no se aplica el método de parsimonia a los STRs del CY La respuesta es muy simple. Los STRs mutan más rápido, de manera que el mismo alelo (por ejemplo 5 repeticiones) se puede generar recurrentemente en diferentes individuos. Se postula que estas secuencias mutan o cambian por un error de la enzima encargada de copiar al ADN (ADN polimerasa) la cual pone repeticiones de más o de menos: un alelo 5 se puede generar a partir de un alelo 4 o de un alelo 6, aunque generalmente se da el primer caso [19]. Sin embargo, el análisis de haplotipos-STRs también aporta valiosa información sobre las relaciones genéticas y peculiaridades de las poblaciones, más aún si se combina con el análisis de marcadores bialélicos [2,15]. Un ejemplo de lo anterior podría ser un estudio de una población hipotética donde se analizaron tres marcadores bialélicos (una sustitución A→G; y una inserción-deleción, +/-) así como 4 STRs, cuyos alelos se representan por números. Se observaron 3 haplogrupos (A/-, A/+, G/+) y una serie de haplotipos-STRs que están "ligados" en una red, donde cada haplotipo se representa por un circulo; su tamaño indica la frecuencia en la población (figura 5).
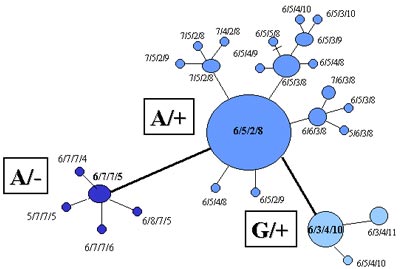
Figura 5. Relación entre haplogrupos y haplotipos STRs del cromosoma Y observados en una población. El tamaño del circulo indica su frecuencia relativa, que permite deducir cuáles son los haplogrupos más antiguos en la población (en este caso el A/+) por su frecuencia y grado de diversificación. Los haplotipos dentro de cada haplogrupo se relacionan por las diferencias en el número de repeticiones de los STRs.¿Qué inferencias poblacionales podríamos hacer
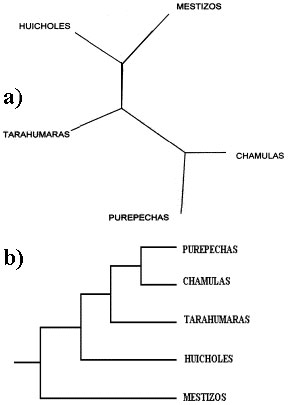
Figura 6. Árboles filogenéticos entre poblaciones mexicanas. El "promedio" de diferencias en los STRs de las poblaciones (frecuencias alélicas) sirve para estimar distancias genéticas, con lo que se elaboran "árboles", que indican la cercanía genética entre poblaciones.En estos estudios de antropología genética, cabe señalar la importancia de integrar la evidencia genética con otras fuentes de información, como la arqueología, la paleontología, la lingüística, la historia, entre otras, para tratar de hacer una reconstrucción lo más fidedigna posible sobre la diversidad genética actual de una población.IV. Conclusiones