HAPLOTIPOS HLA DE RIESGO EN LA ENFERMEDAD CELIACA. ESTUDIO EN POBLACION DE NAVARRA (ESPAÑA)
(especial para SIIC © Derechos reservados)
Coautor
Blanca Hernández Charro*
Licienciada en Ciencias Biológicas. Hospital Virgen del Camino*
Recepción del artículo: 14 de septiembre, 2004
Aprobación: 12 de octubre, 2004
 Conclusión breve
El presente estudio analiza los haplotipos DQB1 y DRB1 en la población celíaca de Navarra (España) y estudia el posible efecto del polimorfismo de la región reguladora 5' del gen HSP70-1 como factor adicional de susceptibilidad
Conclusión breve
El presente estudio analiza los haplotipos DQB1 y DRB1 en la población celíaca de Navarra (España) y estudia el posible efecto del polimorfismo de la región reguladora 5' del gen HSP70-1 como factor adicional de susceptibilidad
 Resumen
Los genes HLA-DQ constituyen los factores genéticos de mayor susceptibilidad para padecer enfermedad celíaca (EC). Pero no todos los genotipos DQ representan el mismo riesgo y, muy probablemente, existen otros genes con importante influencia en la patogénesis de esta enfermedad. El presente estudio analiza los haplotipos DQB1 y DRB1 en la población celíaca de Navarra (España) y estudia el posible efecto del polimorfismo de la región reguladora 5' del gen HSP70-1 como factor adicional de susceptibilidad. Nuestros resultados demuestran que los heterocigotos compuestos DQB1*02-DRB1*03/DQB1*02-DRB1*07 y los homocigotos DQB1*02-DRB1*03 tienen riesgo significativamente superior (OR = 8.23 y OR = 22.0, respectivamente) en relación con los portadores heterocigotos (OR = 1.88), datos que soportan la hipótesis del efecto dosis de DQ2 en el desarrollo de la EC. Además, el alelo HSP70-1*C en heterocigosis y homocigosis, representa un riesgo 2 y 4 veces superior, respectivamente, en los no portadores de los haplotipos de riesgo (DQ2-DR3/DQ2-DR3 y DQ2-DR3/DQ2-DR7). Esta misma tendencia se observa entre los portadores de esos haplotipos, si bien no llega a ser estadísticamente significativa. Estos resultados indican que el alelo C de la HSP70-1 forma parte del haplotipo de riesgo en nuestra población y constituye un factor de susceptibilidad adicional de padecer EC.
Resumen
Los genes HLA-DQ constituyen los factores genéticos de mayor susceptibilidad para padecer enfermedad celíaca (EC). Pero no todos los genotipos DQ representan el mismo riesgo y, muy probablemente, existen otros genes con importante influencia en la patogénesis de esta enfermedad. El presente estudio analiza los haplotipos DQB1 y DRB1 en la población celíaca de Navarra (España) y estudia el posible efecto del polimorfismo de la región reguladora 5' del gen HSP70-1 como factor adicional de susceptibilidad. Nuestros resultados demuestran que los heterocigotos compuestos DQB1*02-DRB1*03/DQB1*02-DRB1*07 y los homocigotos DQB1*02-DRB1*03 tienen riesgo significativamente superior (OR = 8.23 y OR = 22.0, respectivamente) en relación con los portadores heterocigotos (OR = 1.88), datos que soportan la hipótesis del efecto dosis de DQ2 en el desarrollo de la EC. Además, el alelo HSP70-1*C en heterocigosis y homocigosis, representa un riesgo 2 y 4 veces superior, respectivamente, en los no portadores de los haplotipos de riesgo (DQ2-DR3/DQ2-DR3 y DQ2-DR3/DQ2-DR7). Esta misma tendencia se observa entre los portadores de esos haplotipos, si bien no llega a ser estadísticamente significativa. Estos resultados indican que el alelo C de la HSP70-1 forma parte del haplotipo de riesgo en nuestra población y constituye un factor de susceptibilidad adicional de padecer EC.
 Palabras clave
Enfermedad celíaca, haplotipos, HLA-DQ HLA-DR, proteínas de shock térmico, susceptibilidad
Clasificación en siicsalud
Palabras clave
Enfermedad celíaca, haplotipos, HLA-DQ HLA-DR, proteínas de shock térmico, susceptibilidad
Clasificación en siicsalud
 Artículos originales> Expertos del Mundo>
Artículos originales> Expertos del Mundo>
página www.siicsalud.com/des/expertos.php/70152
Especialidades
 Principal: Gastroenterología, Inmunología,
Principal: Gastroenterología, Inmunología,
 Relacionadas: Medicina Interna, Pediatría,
Relacionadas: Medicina Interna, Pediatría,
 Enviar correspondencia a:
Enviar correspondencia a:
María A. Ramos Arroyo. Sección de Genética Médica. Hospital Virgen del Camino. C/ Irunlarrea 4 - 31008 Pamplona. España Ramos-Arroyo, María
HAPLOTIPOS HLA DE RIESGO EN LA ENFERMEDAD CELIACA. ESTUDIO EN POBLACION DE NAVARRA (ESPAÑA)
(especial para SIIC © Derechos reservados)
Artículo completo
Introducción
La enfermedad celíaca (EC) es un trastorno que afecta la mucosa del intestino delgado proximal por intolerancia permanente al gluten. Es la enteropatía alimentaria más frecuente en los países occidentales, con una prevalencia estimada entre 0.3% y 1%. Típicamente se caracteriza por un síndrome de malabsorción con presencia de diarrea crónica, distensión abdominal o escaso desarrollo en peso y estatura (forma clásica), pero puede manifestarse de forma muy diversa con afección musculoesquelética, cutánea, hematológica o neurológica (formas no clásicas) e incluso parecer absolutamente asintomática.1
La EC se puede considerar una enfermedad multifactorial, resultado de la compleja interacción de factores genéticos y ambientales (exposición a proteínas gliadina). La importancia de los factores genéticos de predisposición en la etiología de la EC se basa en diversos pilares de evidencia: a) una mayor prevalencia de la enfermedad (10% a 20%) en familiares de primer grado,2 b) una tasa de concordancia en gemelos monozigóticos en torno del 70%,3 c) una fuerte asociación de la EC con ciertos alelos de sistema HLA (human leucocyte antigen) que codifican moléculas sumamente polimórficas, responsables de la presentación del antígeno peptídico a los receptores de células T en el proceso de activación de éstas.
La región del HLA, localizada en el brazo corto del cromosoma 6 (6p21.3), comprende un segmento de aproximadamente 4 Mb de ADN donde se localizan más de 200 genes. Sobre la base de las características funcionales de estos genes, la región se divide en tres partes, denominadas clases I, II y III. El papel del HLA como factor de susceptibilidad para la aparición de la EC se notificó hace 30 años, implicando inicialmente moléculas presentadoras de antígenos de clase I.4 Posteriormente se observó una asociación con distintos alelos de genes HLA de clase II5-6 (DR3, DR5 y DR7) y fue finalmente en 1983 cuando se demostró la asociación primaria de la EC con los alelos DQ2,7,8 en fuerte desequilibrio de ligamiento con los anteriores. La aparente función de distintos genes de clase III en el proceso de respuesta inmune propició la investigación de su posible papel como factores de susceptibilidad de desarrollar enfermedades como diabetes, EC, artritis reumatoidea, etc. Sin embargo, el fuerte desequilibrio de ligamiento entre los genes de la región HLA dificulta enormemente la determinación del efecto independiente de éstos.
El objetivo de este trabajo es revisar brevemente la información relevante sobre el papel de los genes del sistema HLA de clases II y III en la etiopatogénesis de la EC, y al mismo tiempo ofrecer resultados de nuestras propias investigaciones en la población de Navarra. Nuestro estudio incluye una muestra de 243 pacientes celíacos y 163 controles a los que se realizó análisis de los genes del sistema HLA-DQB1, HLA-DRB1 y del polimorfismo de la región reguladora 5' del gen HSP70-1.9
Genes DQ y DR (HLA clase II)
El principal factor de riesgo de padecer EC identificado hasta el momento es la presencia del heterodímero DQ2 codificado por los alelos DQA1*05 y DQB1*02. Estos alelos pueden estar dispuestos en un mismo cromosoma (haplotipo en cis) con el alelo DRB1*03 (DR3) o en distintos cromosomas (haplotipo en trans) con los alelos DRB1*11 o 12 (DR5) o DRB1*13 (DR6) y DRB1*07 (DR7) (tabla I).
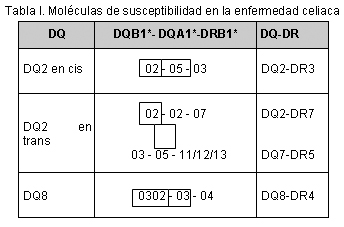
El 90% de los pacientes celíacos son portadores de DQB1*02, en tanto que sólo se observa en 20% a 30%, aproximadamente, de la población general.10 Sin embargo, la combinación de alelos DQB1, DRB1 varía considerablemente en frecuencia según las poblaciones. Así, en el norte de Europa, la gran mayoría de los pacientes celíacos son portadores del haplotipo DQB1*02-DRB1*03 en homocigosis, en tanto que en los países del sur de Europa la combinación en trans, codificada por DRB1*03-DQB1*02/DRB1*07- DQB1*02 o DRB1*11(*12)-DQB1*03/DRB1*07-DQB1*02, es bastante más común.11,12
En la tabla II se detallan las frecuencias de los distintos haplotipos en la población celíaca de Navarra (España). Un 87.2% de los pacientes presentan un genotipo que codifica para la molécula DQ2, y que corresponde en su mayoría a los haplotipos DQ2-DR3 en homocigosis (11.9%) y los heterocigotos compuestos DQ2-DR3/DQ2-DR7 (35%).
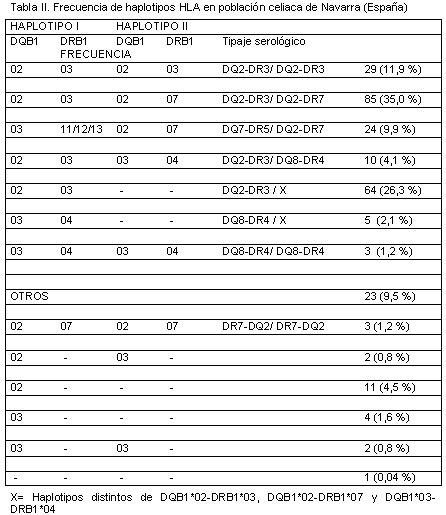
La molécula DQ8, codificada por los alelos DQA1*03-DQB1*0302 asociados a DRB1*04 (DR4) (tabla I), se considera clásicamente el segundo factor en importancia para la susceptibilidad a EC, si bien su efecto parece menos evidente. El genotipo DQB1*03-DRB1*04 está presente en 7.4%, aproximadamente, de los pacientes celíacos de nuestra comunidad, la mitad de los cuales presentan además el haplotipo DQB1*02-DRB1*03. Sólo 9.5% de los casos no presentan ninguno de los haplotipos HLA de riesgo anteriormente mencionados, si bien la mayoría de ellos son portadores del alelo DQB1*02.
El llamativo incremento de los haplotipos DQ2-DR3 en homocigosis y dobles heterocigotos DQ2-DR3/DQ2-DR7 observado en diversas poblaciones sugiere que la susceptibilidad a padecer EC es dependiente del número de copias del heterodímero DQ2,9,13-14 e influye además en el tipo de manifestaciones clínicas y en la edad de comienzo de los síntomas. Diversos estudios avalan hasta el momento este supuesto, en la actualidad se postula que incluso las moléculas DQ2 codificadas por los alelos DQA1*02 y DQB1*02, con DRB1*07, podrían presentar antígenos al gluten, lo que condiciona mayor riesgo de EC a sus portadores.15 Es más, Karell y col.16 sugirieron recientemente que la presencia de una sola de las cadenas de la molécula DQ2 podría ser suficiente para conferir un incremento de riesgo a padecer EC.
La hipótesis del efecto de doble dosis de DQ2 se observa claramente en la población de Navarra. En nuestra muestra de estudio (tabla III) los heterocigotos compuestos DQB1*02-DRB1*03/DQB1*02-DRB1*07 y los homocigotos DQB1*02-DRB1*03 tienen un riesgo significativamente superior (OR:8.2; IC:4.0-17.6; p = 1x10-7 y OR = 22.0; IC: 3.6-902.1; p = 1x10-4, respectivamente) que los portadores heterocigotos DQB1*02-DRB1*03 (OR = 1.9; IC: 1.1-2.2; p = 0.01). Estos resultados son semejantes a los observados en otras poblaciones del sur de Europa,17 donde el genotipo DR3/DR7 es relativamente más frecuente que el DR3/DR3. Sin embargo, difieren de los publicados en la población del norte de Europa (Noruega y Suecia), donde este último genotipo de susceptibilidad es más común. En estas últimas poblaciones, en presencia de DQA1*05-DQB1*02, ambos genotipos confieren riesgo adicional semejante,12,18 lo que indica que el efecto de las moléculas DQ2 codificadas por DQA1*05-DQB1*02(DR3) y DQA1*0201-DQB1*02(DR7) es comparable. La razón de estas diferencias entre poblaciones no es, hoy por hoy, conocida.
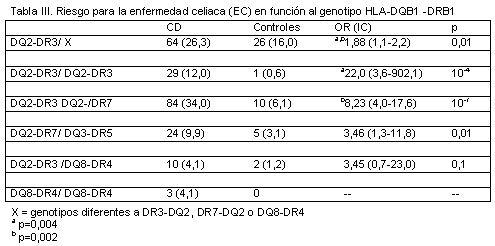
Recientemente se consideró la posibilidad de un efecto semejante respecto de la molécula DQ8.16 En nuestra población, el riesgo para la EC que confiere la presencia de los alelos DQB1*04 y DRB1*03 es poco evidente. Es importante reseñar, sin embargo, que un cuarto de los pacientes celíacos no portadores de DQB1*02-DRB1*03 presentan el haplotipo DQB1*04-DRB1*03. Además, la frecuencia de portadores homocigotos para DQ8 y los de dobles heterocigotos DQ2/DQ8 es superior entre los pacientes celíacos que entre los controles, pero en ninguno de los dos casos la diferencia alcanza significación estadística (tabla III). Es posible que el escaso número de pacientes portadores de dichos haplotipos dificulte en nuestra muestra la consecución de resultados claramente significativos.
Gen HSP70-1 (HLA clase III)
Las proteínas de shock térmico (heat shock [HSP]) son una familia de chaperonas cuya función fundamental es proteger las actividades vitales de la célula ante las agresiones o estrés externo. Pero también parecen tener un papel importante en el desarrollo de la autoinmunidad, en general, y en la patogénesis de las enfermedades autoinmunes en particular.19 Tres de los diversos genes de la familia heat shock se localizan en la región de HLA, circunstancia que hace especialmente interesante su estudio en relación con enfermedades de base inmunológica como la EC.
En 1993, Partaner y col.20 observaron una asociación entre el polimorfismo del gen HSP70-2 y la EC, si bien argumentaban que dicha asociación podría ser debido a desequilibrio de ligamiento entre dicho polimorfismo y el alelo DRB1*03. En 2001 nuestro grupo analizó las variantes alélicas de la region 5' del gen HSP70-1, y observó mayor frecuencia del alelo C en pacientes con EC, incluso en los casos negativos para el alelo DRB1*03, con el que se encuentra en desequilibrio de ligamiento.9 Estos resultados sugerían que el gen HSP70-1 podría ser un factor de susceptibilidad adicional en la aparición de la EC. El incremento de la muestra de estudio, tanto de casos como de controles nos permite reevaluar dicho efecto y confirmar los resultados previamente publicados al respecto.
La frecuencia del alelo C de la HSP70-1 en nuestra población es significativamente superior en la población celíaca (60.6%) en relación con los controles (37.5%). Esta misma tendencia se observa independientemente de la presencia de los alelos DRB1*03 y DQB1*02, con los que se encuentra en desequilibrio de ligamiento, si bien el incremento no alcanza significación estadística (tabla IV). Con el fin de intentar definir este posible efecto del alelo HSP70-1*C analizamos el riesgo de los distintos genotipos considerados de susceptibilidad para la EC en nuestra población (DQB1*02-DRB1*03/DQB1*02-DRB1*03 o DQB1*02-DRB1*03/DQB1*02-DRB1*07), en relación con el genotipo de referencia, definido como aquel no portador de los mismos. Como se observa en la tabla V, la presencia del alelo C, tanto en heterocigosis como en homocigosis, representa un riesgo de padecer EC de 2 y 4 veces superior, respectivamente, en los no portadores de los haplotipos de riesgo DQ y DR. Del mismo modo, la presencia del alelo HSP70-1*C confiere un riesgo adicional de padecer EC en los portadores de los haplotipos de susceptibilidad, es especialmente llamativo en los portadores homocigotos (OR = 47.7; CI: 10.9-419.3); p = 10-7). A pesar de que este incremento de riesgo no alcanza en nuestra muestra significación estadística, probablemente debido al pequeño tamaño muestral, los resultados conjuntos del análisis sugieren que el alelo C de la HSP70-1 forma parte del haplotipo de riesgo en nuestra población y constituye un factor de susceptibilidad adicional para padecer EC.


El posible papel de la HSP70-1 en el complejo mecanismo etiopatogénico de la EC es hoy por hoy desconocido. Diversos estudios funcionales demostraron que la reacción en cascada que da lugar a la EC se inicia en el momento en que el gluten traspasa el epitelio del intestino y llega a la lámina propia. Allí, las moléculas HLA (DQ2 y DQ8) de las células presentadoras de antígenos, reconocen esas proteínas y las presentan a los receptores de células T CD4+, iniciándose la activación de los linfocitos T citotóxicos y la producción de citoquinas, así como el desarrollo de células B que producen anticuerpos contra el gluten y transglutaminasa. Como consecuencia, se produce hiperplasia de las células crípticas y la lesión de la mucosa intestinal. Las proteínas HSP son mediadoras en el proceso de plegamiento proteico y podrían jugar un papel importante en el procesamiento y presentación de antígenos.21,22 Es posible, por ello, que algunos de los polimorfismos de los genes HSP70 puedan modificar el complejo HLA-péptido de presentación a las células T receptoras, constituyendo así factores de susceptibilidad o protección para la EC en condiciones de estrés celular.
Es también posible, por otro lado, que el alelo HSP70-1*C sea parte de un haplotipo conservado del sistema HLA que a su vez incluya otros genes de susceptibilidad a desarrollar EC. La región genómica HLA alberga numerosos genes de interés por su papel en el proceso de autoinmunidad y su expresión en el epitelio intestinal, como el TNF2, MICA o MICB, entre otros. Sin embargo el fuerte desequilibrio de ligamiento que presentan estos genes con DQB1 y DRB1 dificulta la obtención de resultados claros respecto de su función como genes de susceptibilidad para desarrollar EC.23-26 A pesar de ello, la presencia de factores de riesgo dentro de la región HLA, además de la presencia de DQ2, parece actualmente evidente, por lo que la investigación en este campo continúa siendo de gran relevancia.
Consideraciones finales
El papel fundamental de los genes DQ como factores de susceptibilidad para padecer EC es hoy incuestionable. Sin embargo, quedan aún numerosos aspectos sin explicar que sin duda requieren investigación en mayor profundidad. ¿Cuál es el verdadero fundamento del efecto dosis del gen DQB1*02 ¿Existe ese mismo efecto con respecto a la molécula DQ8 ¿Por qué el riesgo asociado al genotipo DR3/DR3 es semejante al conferido por DR3/DR7 entre la población del norte de Europa y diferente en Europa del sur ¿Cuál es el verdadero significado de la aparente asociación de otros genes de las regiones I (MICB) y III (MICA, TNF, HSP70-1) del sistema HLA, descritas en diversas poblaciones ¿Existen otros genes de susceptibilidad fuera de la región HLA La resolución de estas dudas y el conocimiento preciso del mecanismo de acción de éstos y otros genes requerirá tanto la continuación de los estudios genéticos como los avances en la investigación de sus funciones a nivel tisular.
Los autores no manifiestan conflictos.
 Bibliografía del artículo
Bibliografía del artículo
- Maki M, Collin P. Coeliac disease. Lancet 1997 Jun 14;349(9067):1755-9.
- Maki M, Holm K, Lipsanen V y col. Serological markers and HLA genes among healthy first-degree relatives of patients with coeliac disease. Lancet 1991 Nov 30;338(8779):1350-3.
- Polanco I, Biermond I, Van Leeuwen A y col. Gluten sensitive enteheropathy in Spain: genetic and enviromental factors. In: The genetics of coeliac disease. Ed McConnel RB. MTP Press, Lancaster, 1981, pp:211-231.
- Stokes PL, Asquith P, Holmes GK y col. Histocompatibility antigens associated with adult coeliac disease Lancet 1972 Jul 22; 2(7769):162-4.
- Keuning JJ, Pena AS, Van Leeuwen A y col. HLA-DW3 associated with coeliac disease. Lancet 1976 Mar 6; 1(7958):506-8.
- Mearin ML, Biemond I, Pena AS y col. HLA-DR phenotypes in Spanish coeliac children: their contribution to the understanding of the genetics of the disease. Gut 1983 Jun; 24(6):532-7.
- Tosi R, Vismara D, Tanigaki N y col. Evidence that celiac disease is primarily associated with a DC locus allelic specificity Clin Immunol Immunopathol. 1983 Sep; 28(3):395-404.
- Sollid LM, Markussen G, Ek J y col. Evidence for a primary association of celiac disease to a particular HLA-DQ alpha/beta heterodimer. J Exp Med. 1989 Jan 1; 169(1):345-50.
- Ramos Arroyo MA, Feijoo E, Sanchez Valverde F y col. Heat-shock protein 70-1 and HLA class II gene polymorphisms associated with celiac disease susceptibility in Navarra (Spain). Hum Immunol. 2001 Aug; 62(8):821-5.
- Herrera M, Theiler G, Augustovski F y col. Molecular characterization of HLA class II genes in celiac disease patients of Latin American Caucasian origin. Tissue Antigens 1994 Feb; 43(2):83-7.
- Tighe MR, Hall MA, Barbado M y col. HLA class II alleles associated with celiac disease susceptibility in a southern European population Tissue Antigens. 1992 Aug; 40(2):90-7.
- Ploski R, Ascher H, Sollid LM. HLA genotypes and the increased incidence of coeliac disease in Sweden. Scand J Gastroenterol. 1996 Nov; 31(11):1092-7.
- Fernandez Arquero M, Figueredo MA, Maluenda C y col. HLA-linked genes acting as additive susceptibility factors in celiac disease. Hum Immunol 1995 Apr; 42(4):295-300.
- Ploski R, Ek J, Thorsby E y col. On the HLA-DQ(alpha 1*0501, beta 1*0201)-associated susceptibility in celiac disease: a possible gene dosage effect of DQB1*0201. Tissue Antigens 1993 Apr;41(4):173-7.
- Vader W, Stepniak D, Kooy Y y col. The HLA-DQ2 gene dose effect in celiac disease is directly related to the magnitude and breadth of gluten-specific T cell responses. Proc Natl Acad Sci U S A. 2003 Oct 14; 100(21):12390-5.
- Karell K, Louka AS, Moodie SJ y col. European Genetics Cluster on Celiac Disease. HLA types in celiac disease patients not carrying the DQA1*05-DQB1*02 (DQ2) heterodimer: results from the European Genetics Cluster on Celiac Disease. Hum Immunol. 2003 Apr; 64(4):469-77.
- Meddeb-Garnaoui A, Zeliszewski D, Mougenot JF y col. Reevaluation of the relative risk for susceptibility to celiac disease of HLA-DRB1, -DQA1, -DQB1, -DPB1, and -TAP2 alleles in a French population. Hum Immunol 1995 Jul; 43(3):190-9.
- Louka AS, Nilsson S, Olsson M y col. HLA in coeliac disease families: a novel test of risk modification by the 'other' haplotype when at least one DQA1*05-DQB1*02 haplotype is carried. Tissue Antigens. 2002 Aug; 60(2):147-54.
- Van Buskirk AM, DeNagel DC, Guagliardi LE y col. Cellular and subcellular distribution of PBP72/74, a peptide-binding protein that plays a role in antigen processing. J Immunol. 1991 Jan 15;146(2):500-6.
- Partanen J, Milner C, Campbell RD y col. HLA-linked heat-shock protein 70 (HSP70-2) gene polymorphism and celiac disease. Tissue Antigens. 1993 Jan;41(1):15-9.
- Gruen JR, Weismann SM. Human MHC class III and IV genes and disease associatios. Frontiers in Biosceince 2001; 6:960-972.
- Georgepoulus C, Welch WJ: Role of the major heat shock proteins as molecular chaperones. Annu Rev Cell Biol 1993; 9:601-03.
- McManus R, Moloney M, Borton y col. Association of celiac disease with microsatellite polymorphisms close to the tumor necrosis factor genes. Hum Inmunol 1996; 45:24-31.
- De la Concha EG, Fernández Arquero M, Vigil P y col. Celiac disease and TNF promoter polymorphisms. Hum Inmunol 2000; 61:513-17.
- Bolognesi E, Kareti K, Percopo y col. Additional factor in some HLA DR3/DQ2 haplotypes confers a fourth increased genetic risk of celiac disease. Tissue Antigens 2003; 61:308-316.
- Louka AS, Sollid LM. HLA in coeliac disease: unravelling the complex genetics of a complex disorder. Tissue Antigens. 2003 Feb; 61(2):105-17.
©
Está
expresamente prohibida la redistribución y la redifusión de todo o parte de los
contenidos de la Sociedad Iberoamericana de Información Científica (SIIC) S.A. sin
previo y expreso consentimiento de SIIC






 Artículos originales> Expertos del Mundo>
Artículos originales> Expertos del Mundo> Enviar correspondencia a:
Enviar correspondencia a:



