La combinación de métodos de redes sociales y epidemiología molecular comenzó a utilizarse recientemente en la investigación de enfermedades infecciosas. La utilización conjunta de estas técnicas permite identificar y descifrar patrones de enfermedad en agrupaciones complejas de seres humanos; así, la primera resulta útil para evaluar cómo se transmite la infección, mientras que la segunda se aplica a las relaciones y conductas que posibilitan esta transmisión. A partir del año 2000 comenzamos un estudio en el cual se combinaron por primera vez los métodos de redes sociales con la epidemiología molecular a fin de avanzar en nuestro conocimiento sobre la epidemiología del virus de la hepatitis C (VHC) entre usuarios de drogas intravenosas (UDI), para lo cual reclutamos una cohorte de 199 UDI, de los cuales 138 tenían ARN positivo para VHC, en una zona utilizada para consumo de drogas en Melbourne, Australia. Estudiamos muestras de sangre obtenidas de los participantes, al tiempo que se evaluaron datos informados por ellos, por medio del análisis de redes sociales y análisis moleculares filogenéticos de dos regiones del genoma del VHC. Los hallazgos de este estudio (denominado en lo sucesivo como Redes I) fueron publicados en varios artículos entre 2004 y 2005,1-4 sus principales resultados se detallan a continuación:
- hubo una baja correlación entre las relaciones identificadas por técnicas de redes sociales y las que se delinearon por datos filogenéticos;1
- la distancia social (número de vínculos que separan a los UDI) mostró escasa relación con la distancia genética entre secuencias virales;1
- el cambio del fenotipo del VHC entre UDI se presenta al menos tan frecuentemente como las infecciones en individuos no infectados previamente, mientras que las tasas de incidencia del VHC calculadas tradicionalmente representan una minoría en la transmisión real del VHC entre UDI;2
- algunos UDI pueden ejercer conductas de alto riesgo para infección por VHC, con frecuencia similar y durante períodos comparables a los de otros UDI, sin presentar marcadores de infección:3
- las infecciones por genotipos mixtos de VHC se presentan en un subgrupo significativo de UDI y no se detectan con facilidad por ensayos de sondas alineadas (line-probe assay [LiPA], Bayer Diagnostics), que es el método más común de genotipificación.4
Aunque todos los resultados son relevantes, para mantener la claridad y brevedad del presente artículo nos ocuparemos principalmente de los dos primeros, que pueden describirse simultáneamente como mediciones de la correspondencia entre los datos de redes sociales y redes moleculares. Además de explorar los motivos por los que se obtuvieron estos resultados en Redes I, describiremos nuestra investigación actual (Redes II) y
mencionaremos algunos de los datos preliminares obtenidos; asimismo, efectuaremos una breve revisión sobre las nuevas investigaciones en este tema, conocidas luego de la publicación de nuestros datos.
Correlación entre redes sociales y datos moleculares
En Redes I se solicitó a los UDI que propusieran o presentaran a nuestros trabajadores de campo a los integrantes de su red, que eran las personas con quienes se inyectaban drogas al mismo tiempo y en igual ubicación, al menos una vez en los 6 meses previos. Nuestros registros reunieron 577 relaciones diádicas o de pares que se inyectan y 66 pares de UDI con infecciones identificadas como posiblemente relacionadas filogenéticamente. Entre los pares de UDI con infecciones por VHC genéticamente relacionadas, sólo el 18.2% estaban socialmente vinculados, mientras que únicamente un 3.4% de los sujetos con ARN positivo vinculados socialmente tenían infecciones relacionadas. El bajo nivel de correlación entre nuestros datos filogenéticos y de redes sociales pudo deberse a una combinación de los siguientes motivos:
- Por parte de la red social, se presenta una situación de captura incompleta de un cuadro complejo;5 por lo que nuestra habilidad de rastrear patrones de infección por VHC a través de la red social de UDI se vio disminuida por las limitaciones prácticas de encontrar y entrevistar a UDI específicos en un escenario callejero rápidamente cambiante.
- La naturaleza epidémica del VHC entre UDI dificulta la reconstrucción de patrones de infección; además, el VHC mantiene una elevada prevalencia entre UDI en Australia, ya que el 61% de los participantes en la encuesta del programa nacional de agujas y jeringas de Australia, tuvieron pruebas para anti-VHC positivas en 2005.6 Las mediciones recientes de incidencia entre UDI jóvenes o que se inician mostraron un 133% por año entre australianos que se han inyectado durante menos de 1 año7 y 41.8% por año entre UDI británicos menores de 30 años, o que se han inyectado por 6 años o menos.8 Los participantes en Redes I se habían estado inyectando durante más de 10 años en promedio y la prevalencia de anticuerpos contra VHC fue cercana al 85%; así, en la mayor parte de los casos, la primera exposición al VHC probablemente había ocurrido años antes de la entrevista, más allá de que para la compleja información de la red social pudo existir algún período mal recordado.
- Nos vimos obligados a confiar en lo informado por nuestros participantes, con respecto al uso de drogas inyectables en los 6 meses previos, a fin de asegurar una recuperación razonable de la información. También trabajamos sobre la base de la presunción de que así se maximizaban nuestras probabilidades de reclutar integrantes de la red de UDI; ya que era probable que las personas con las cuales nuestros entrevistados se hubieran inyectado recientemente podrían encontrarse más cerca que otros que hubieran compartido esta práctica en momentos anteriores. De hecho, estos métodos limitaron el volumen potencial de integrantes de la red, aunque probablemente permanecieron aquellos UDI en los cuales era más probable detectar infecciones relacionadas, debido a una menor cantidad de divergencias entre las secuencias del VHC.
- Las infecciones con genotipos múltiples del VHC no son bien identificadas por el ensayo de sondas alineadas (LiPA),9,10 pero pueden constituir una fracción importante de todas las infecciones.4 A lo largo del tiempo, el genotipo dominante detectado por LiPA puede variar11 en personas infectadas con más de un genotipo del VHC; por lo que estas personas, que podrían haber sido reconocidas por presentar secuencias relacionadas del VHC, ya no serían identificadas como tales.
Redes II
La consecuencia práctica de haber hallado en Redes I una mala correlación entre nuestros datos sociales y moleculares, además del interesante descubrimiento de UDI expuestos a alto riesgo durante mucho tiempo, pero no infectados, fueron factores determinantes en nuestra decisión de realizar un segundo estudio, basado en los resultados del primero, pero evitando la mayor cantidad posible de errores. La recolección de datos para Redes II se llevó a cabo entre julio de 2005 y agosto de 2006, y en ese período trabajaron 5 investigadores externos que entrevistaron a los participantes y tomaron al menos una muestra de sangre venosa de 319 sujetos (y al menos 2 muestras de 173) que eran UDI actuales. Como se mencionó anteriormente, el reclutamiento se realizó en tres locaciones de la ciudad de Melbourne (capital del estado de Victoria, Australia; población en 2004: 3 592 975 habitantes),12 cada una de las cuales aloja un escenario de consumo de drogas ilícitas, en las que existen sitios fijos dedicados al programa nacional de agujas y jeringas. Las muestras de sangre para la realización de análisis para VHC, VIH y virus de la hepatitis B fueron obtenidas luego de dar un asesoramiento completo; los resultados se entregaron en privado, también junto con un asesoramiento completo. Se solicitó a los participantes que propusieran o presentaran a los integrantes de su grupo o red (personas con las cuales se hubieran inyectado drogas, en igual ocasión y lugar, con el mismo criterio utilizado para Redes I) para entrevistarlos. La recolección de datos se vio facilitada por el uso de computadoras de mano, sincronizadas con una base de datos central; la mayoría de las entrevistas fueron conducidas, mientras que la extracción de las muestras de sangre se realizó en una camioneta, que también fue utilizada para el transporte de los trabajadores de campo, desde y hacia los sitios de reclutamiento, ampliamente distanciados. Las principales diferencias entre Redes II y Redes I son las siguientes.
Diseño longitudinal en vez de transversal
Redes II es un estudio prospectivo longitudinal, en el cual los UDI serán entrevistados hasta nueve veces durante un período de 2 años, con repetición de las entrevistas aproximadamente cada 3 meses, lo que brinda la posibilidad de observar el espectro completo de la dinámica de la infección por VHC (infección incidental, remoción del virus, de los anticuerpos o de ambos, desarrollo de cronicidad y sobreinfección). Las entrevistas reiteradas también permiten observar la evolución de las redes, e incrementan nuestra capacidad para detectar nuevas infecciones por VHC; además de rastrear la transmisión, y detectar participantes que adquieren nuevos compañeros de inyección de drogas con ARN positivo para VHC.
Menor énfasis en la densidad de las redes
En Redes II se utilizan tres sitios de reclutamiento ampliamente distanciados entre sí, contra un sitio único en Redes I; además, mientras que en Redes I los trabajadores de campo sólo podían reclutar personas presentadas como integrantes de la red por los entrevistados, en Redes II esa regla fue flexibilizada. El efecto de esos cambios fue una menor densidad y complejidad de las redes, lo que simplificó las relaciones y facilitó el reconocimiento de los patrones de infección; las figuras 1 y 2 muestran cuán diferentes son las estructuras de las redes de relaciones entre usuarios de inyectables, que se produjeron en Redes I y Redes II hasta ahora. La dispersión geográfica de los sitios de reclutamiento tuvo como beneficio adicional la posibilidad de aplicar la epidemiología molecular “clásica”, para demostrar cómo las infecciones por VHC son transportadas desde un lugar de la ciudad a otro; así, la investigación de 77 secuencias de la región central del genotipo 1a (derivadas de especímenes de los primeros UDI) nos permitió documentar infecciones por VHC relacionadas genéticamente, en dos pares de UDI que vivían a 40 y 50 km de distancia.13

Figura 1. Redes I.
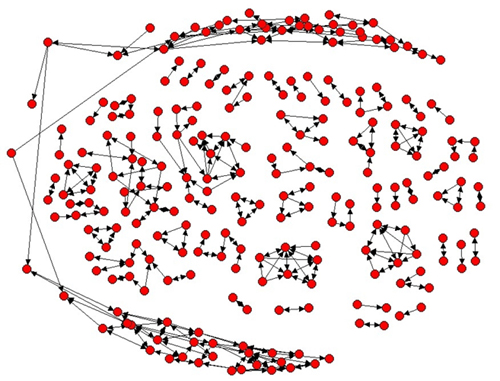
Figura 2. Redes II.
Enfasis en UDI jóvenes o de inicio reciente
En Redes I los UDI fueron reclutados basándose sólo en su identificación como miembros de las redes por otros entrevistados; mientras que en Redes II, las personas menores de 25 años que se habían estado inyectando por menos de tres años fueron reclutados en forma preferencial (denominados participantes primarios, con seguimiento longitudinal). Por su parte, los participantes secundarios no se incluyeron en el seguimiento, salvo que se hubiera probado que no habían estado expuestos al VHC. Se intentó incluir UDI jóvenes o de inicio reciente, a fin de obtener un número suficiente de UDI sin infección por VHC para poder efectuar comparaciones estadísticas con los individuos expuestos (algo que no fue posible en Redes I, ya que se reclutaron sólo 21 UDI no expuestos); además de permitirnos medir en forma más exacta la incidencia, e incluir UDI expuestos al VHC con historias de infección más corta y de menor complejidad, comparadas con las de Redes I. De esta manera, pudimos mejorar nuestra capacidad para detectar infecciones relacionadas por epidemiología molecular; estrategia que parece funcionar bien, ya que logramos identificar 25 casos de infecciones nuevas entre 105 UDI inicialmente negativos para anti-VHC, lo que indica una incidencia de 37.6% anual (95% IC 21.9-64.8). El promedio de uso de drogas inyectables de los individuos seguidos en Redes II fue de 7.8 años (95% IC 7.1-8.4), que resulta significativamente menor que el de Redes I, de 10.2 años (95% IC 9.2-11.3).
Inclusión de la inmunología
Encontramos un subgrupo de participantes de Redes I, que pese a encontrarse en situación de alto riesgo para infección por VHC, no presentaba evidencias de exposición,3 lo que coincide con informes previos de otros autores14 que plantearon como hipótesis que algunos individuos poseen o pueden adquirir una función inmune que los proteja de la infección por VHC, o bien les permita remover la infección rápidamente, sin producción detectable de anticuerpos. Nosotros comenzamos a colaborar informalmente con los principales inmunólogos australianos hacia el final del estudio Redes I; mientras que la colaboración formal se inició en Redes II. Así, contamos con una gran reserva de muestras de sangre venosa (hasta 50 ml) obtenidas de UDI persistentemente negativos, seroconvertidos y UDI sobreinfectados con ARN positivo persistente; la cual brinda el material crudo para análisis que permiten el estudio de la inmunidad para VHC innata y adaptativa, y facilitan la creación de posibles vacunas.
Hasta el momento, las respuestas de células T específicas para VHC se identificaron en sujetos seronegativos y ARN negativos, contra determinantes de células T CD8+, los cuales también son reconocidos por participantes infectados por VHC, ARN positivos, o ambos. Esto confirma que los UDI persistentemente negativos han estado efectivamente expuestos al VHC, pero muestran una respuesta inmune más poderosa que les permiten frustrar tanto la viremia crónica como la producción de anticuerpos.
Análisis de curvas de fusión de PCR en tiempo real para discriminación del genotipo
En el estudio Redes I realizamos pruebas en una pequeña parte de la cohorte con análisis de movilidad heterodúplex (heteroduplex mobility assay [HMA]),4 con lo que detectamos que 3.6% de las muestras de UDI con ARN positivo para VHC contenían más de un genotipo viral. Nuestra experiencia subsecuente nos hizo comprender que la cifra real es varias veces mayor y supera probablemente el valor de 7.6% informado por Schroter y col.15 Mientras que la técnica HMA es útil para detectar genotipos múltiples, no permite discriminar fácilmente entre ellos; por lo tanto, nosotros desarrollamos una prueba que utiliza la curva de fusión en tiempo real, que incrementa significativamente la posibilidad de identificar los genotipos presentes en una infección mixta. Brevemente, la discriminación entre genotipos resulta posible por la implementación de una sonda específica para los productos de PCR de un genotipo, pero es lo suficientemente complementaria como para ligarse a otros; así, en el análisis de las curvas de fusión, la sonda se disocia de los productos de PCR menos específicos a una temperatura característica, menor a la de los específicos, que depende del genotipo.
Investigaciones nuevas luego de Redes I
Podría decirse que la principal razón por la cual los resultados de Redes I mostraron mala correspondencia entre los datos de redes sociales y secuencias del VHC, fue el hecho de que los datos utilizados provenían de UDI con antecedentes de infección por VHC complejos y de larga data. En el diseño de Redes II se buscó enmendar este problema, al enfocarse en usuarios de drogas inyectables más jóvenes o de inicio reciente; estrategia que parece ser exitosa, según se mencionó previamente. Uno de nuestros objetivos al realizar Redes II fue utilizar una combinación de análisis de redes sociales y epidemiología molecular, para comparar directamente la epidemiología social y molecular de pares de UDI que incluían al menos un episodio de infección por VHC. Estas personas, por definición, habrían adquirido la infección en forma reciente de otro UDI que, debido a que el evento de transmisión era reciente, probablemente continuara formando parte de la red social “infectante”. Así, podríamos esperar que los vínculos sociales y los datos moleculares muestren una correspondencia mucho mayor que la observada en Redes I, en el cual la cohorte de UDI presentaba infecciones por VHC prevalentes, más que episódicas.
En la realización de un estudio retrospectivo de casos y controles, acerca de UDI con infecciones por VHC incidentales (n = 17) y sin evidencia de exposición (n = 42), Brewer y col.16 presentaron datos que confirman la lógica del abordaje bosquejado más arriba. En tres de cuatro pares de infecciones encontradas por estos autores, existían relaciones estrechas, y los UDI involucrados habían mencionado cada uno al otro como integrante de la red de usuarios de inyectables; así, aunque el número de casos es pequeño, el concepto mantiene su validez. Resultó llamativo que en el trabajo de Brewer y col.16 no hayan encontrado correspondencia entre la distancia genética y de red entre sujetos con infecciones nuevas, ya que esto sugiere que la distancia genética entre secuencias del VHC probablemente no sea representativa de la distancia social entre los huéspedes. Nosotros esperamos poder reproducir estos resultados con números superiores de casos nuevos y de controles, antes de la probable finalización de la recolección de datos para Redes II, a mediados de 2007.
Ha surgido mucha información nueva respecto de nuestra comprensión de los aspectos inmunológicos de la infección por VHC y se lograron avances importantes en el trabajo de preparación para la producción de una vacuna. Prince y col.17 mostraron que en chimpancés recuperados de infecciones previas por genotipo 1a o 1b se produjeron infecciones crónicas luego de una prueba de provocación con genotipos heterólogos, lo que sugiere que se requerirán epitopes de varios genotipos para obtener una vacuna contra el VHC ampliamente efectiva. Encke y col.18 partieron de un enfoque diferente, en el que vacunaron ratones con células dendríticas con el core del VHC y detectaron importantes respuestas inmunes, tanto de tipo celular como humoral. Los hallazgos de Folgori y col.,19 quienes indujeron protección de genotipo cruzado para hepatitis aguda en chimpancés mediante una vacuna genética de células T para VHC, son prometedores. Así demostraron que se puede lograr una protección efectiva contra varias cepas del VHC por medio de la estimulación de la inmunidad celular únicamente.
La investigación inmunológica que realizamos en Redes II estuvo concentrada en mejorar nuestros conocimientos acerca de las respuestas efectivas de las células T (determinantes peptídicos específicos, frecuencias de células T, fenotipos funcionales y el repertorio de células T) que conducen a la resistencia al VHC y su remoción; además de identificar determinantes de células T reconocidos por los diferentes genotipos virales. Como ya se mencionó, actualmente existe una evidencia considerable de que las respuestas de inmunidad celular son esenciales en la resolución espontánea de la hepatitis aguda por virus C y para la protección a largo plazo frente a la infección persistente; en consecuencia, las estrategias dirigidas a potenciar las respuestas de células T específicas para VHC pueden cumplir un importante papel en la creación de vacunas. Sin embargo, aunque existen seis genotipos del VHC y numerosos subtipos, la mayor parte de los trabajos publicados se concentraron en el genotipo 1, que corresponde a un 60% de las infecciones en UDI australianos,20,21 mientras que unos pocos estudios estuvieron dirigidos al genotipo 3, el siguiente en frecuencia.20 Nosotros estamos trabajando con cohortes protegidas de UDI (negativos para anti-VHC, para PCR y PCR-negativos con anti-VHC positivo), a fin de investigar la correlación entre la inmunidad protectora en la infección por VHC y facilitar el diseño de una subunidad efectiva como vacuna y estrategia inmunoterapéutica. Identificamos nuevos determinantes específicos para VHC en células T CD8+, desde el genotipo 3 restringido para un amplio número de alelos de HLA. Combinado con las secuencias del epitope de linfocitos T citotóxicos (CTL) (publicadas en http://hcv.lanl.gov/content/immuno/tables/ctl_summary.html) del genotipo 1 del VHC, el gran repertorio de péptidos asegura un amplio intervalo de estudio para UDI que muestran alta prevalencia de genotipo 3 y que en muchos casos son portadores de más de un genotipo viral. Nos proponemos efectuar un riguroso análisis longitudinal del repertorio de células T en UDI con resultados clínicos diferentes; principalmente resistencia viral (anti-VHC y PCR negativos), clearance o depuración (anti-VHC positivo con ARN negativo), o persistencia (anti-VHC y PCR positivos). Esta comparación revelará la especificidad y frecuencia de las células T CD8+ específicas para VHC asociadas con protección y permitirá conocer su composición clonal. Asimismo, los UDI infectados con genotipos del VHC diferentes del 1 (genotipos 2, 3 y 6) serán sometidos a tamizaje en el panel completo de péptidos del VHC a fin de identificar determinantes de células T con reconocimiento cruzado entre genotipos, útiles para la creación de futuras vacunas.
Conclusiones
Por medio de Redes II estamos intentando proseguir con la utilización de métodos de redes sociales y epidemiología molecular para mejorar nuestra comprensión acerca de la epidemiología del VHC, su virología y sus interacciones. Debido a la posibilidad de mapear los patrones de transmisión del virus entre redes y de caracterizar en términos biológicos y de conducta, tanto a la fuente de infección como al huésped, pensamos que este enfoque ofrece una excelente base para las investigaciones sobre la inmunología del VHC que puede resultar útil para facilitar el proceso de creación de una vacuna.
BIBLIOGRAFÍA
1. Aitken CK, McCaw R, Bowden S, Tracy SL, Kelsall J, Higgs P, Kerger M, Nguyen H, Crofts N. Molecular epidemiology of the hepatitis C virus in a social network of injecting drug users. Journal of Infectious Diseases 190(9):1586-1595, 2004.
2. Aitken CK, Bowden S, McCaw R, Jardine D, Hellard M, Higgs P, Nguyen O, Crofts N. Change in hepatitis C virus genotype in injecting drug users. Journal of Medical Virology 74 (4):543-545, 2004.
3. Aitken CK, Bowden S, Hellard M, Crofts N. Indications of immune protection from hepatitis C infection. Journal of Urban Health 81(1):58-60, 2004.
4. Bowden S, McCaw R, White P, Crofts N, Aitken CK, Detection of multiple hepatitis C virus genotypes in a cohort of injecting drug users. Journal of Viral Hepatitis 12:322-324, 2005.
5. Rothenberg R, Baldwin J, Trotter R, Muth SQ, The risk environment for HIV transmission: results from the Atlanta and Flagstaff network studies. J Urban Health 78(3):419-432, 2001.
6. National Centre in HIV Epidemiology and Clinical Research. Australian NSP Survey National Data Report 2001-2005. National Centre in HIV Epidemiology and Clinical Research, The University of New South Wales, Sydney, NSW, 2006.
7. Maher L, Jalaludin B, Chant KG, Jayasuriya R, Sladden T, Kaldor JM, Sargent PL. Incidence and risk factors for hepatitis C seroconversion in injecting drug users in Australia. Addiction 101(10):1499-508, 2006.
8. Judd A, Hickman M, Jones S, McDonald T, Parry JV, Stimson GV, Hall AJ. Incidence of hepatitis C virus and HIV among new injecting drug users in London: prospective cohort study. British Medical Journal 330(7481):24-5, 2005.
9. Hu YW, Balaskas E, Furione M, Yen PH, Kessler G, Scalia V, Chui L, Sher G, Comparison and application of a novel genotyping method, semiautomated primer-specific and mispair extension analysis, and four other genotyping assays for detection of hepatitis C virus mixed-genotype infections. J Clin Microbiol 38(8):2807-2813, 2000.
10. Giannini C, Giannelli F, Monti M, Careccia G, Marrocchi ME, Laffi G, Gentilini P, Zignego AL. Prevalence of mixed infection by different hepatitis C virus genotypes in patients with hepatitis C virus-related chronic liver disease. J Lab Clin Med 134(1):68-73, 1999.
11. Laskus T, Wang LF, Radkowski M, Nowicki M, Wilkinson J, Rakela J. Exposure of hepatitis C virus-negative recipients to > or = 2 infected blood donors. Journal of Infectious Diseases 183(4):666-669, 2001.
12. Australian Bureau of Statistics, National Regional Profile: Melbourne (Statistical Division), 2006. www.abs.gov.au/AUSSTATS/abs@.nsf/Latestproducts/205Population/People12000-2004?opendocument&tabname=Summary&prodno=205&issue=2000-2004&num=&view= (last viewed 2 October 2006).
13. McCaw R, Hellard M, Aitken C, Tray L, Revill P, Bharadwaj M, Bowden S. Virological and epidemiological factors influencing hepatitis C infection in injecting drug users. 13th International Meeting on Hepatitis C Virus & Related Viruses 27-31 August 2006.
14. Mehta SH, Cox A, Hoover DR, et al, Protection against persistence of hepatitis C. Lancet 359(9316):1478-83, 2002.
15. Schroter M, Feucht HH, Zollner B, Schafer P, Laufs R. Multiple infections with different HCV genotypes: prevalence and clinical impact. J Clin Virol 27(2):200-4, 2003.
16. Brewer DD, Hagan H, Sullivan DG, Muth SQ, Hough ES, Feuerborn NA, Gretch DR. Social structural and behavioral underpinnings of hyperendemic hepatitis C virus transmission in drug injectors. Journal of Infectious Diseases 194(6):764-72, 2006.
17. Prince AM, Brotman B, Lee DH, Pfahler W, Tricoche N, Andrus L, Shata MT. Protection against chronic hepatitis C virus infection after rechallenge with homologous, but not heterologous, genotypes in a chimpanzee model. Journal of Infectious Diseases 192(10):1701-9, 2005.
18. Encke J, Findeklee J, Geib J, Pfaff E, Stremmel W. Prophylactic and therapeutic vaccination with dendritic cells against hepatitis C virus infection. Clin Exp Immunol 142(2):362-9, 2005.
19. Folgori A, Capone S, Ruggeri L, Meola A, Sporeno E, Ercole BB, Pezzanera M, Tafi R, Arcuri M, Fattori E, Lahm A, Luzzago A, Vitelli A, Colloca S, Cortese R, Nicosia A. A T-cell HCV vaccine eliciting effective immunity against heterologous virus challenge in chimpanzees. Nauret Medicine 12(2):190-7, 2006.
20. Freeman AJ, Zekry A, Whybin LR, Harvey CE, van Beek IA, de Kantzow SL, Rawlinson WD, Boughton CR, Robertson PW, Marinos G, Lloyd AR. Hepatitis C prevalence among Australian injecting drug users in the 1970s and profiles of virus genotypes in the 1970s and 1990s. Med J Aust 172(12):588-91, 2000.
21. McCaw R, Moaven L, Locarnini SA, Bowden DS, Hepatitis C virus genotypes in Australia. J Viral Hepat 4(5):351-7, 1997.
|


![]()
